Prérequis
Les participants devront apporter :
- leur propre matériel informatique (PC portable, système d'exploitation indifférent) qui leur permettra de se connecter à l'infrastructure,
- un jeu de données, en accord avec leur tuteur, qu'ils analyseront lors les séances "Tutorat" en fin de semaine (des jeux de référence en MS et RMN seront également disponibles sur l'infrastructure).
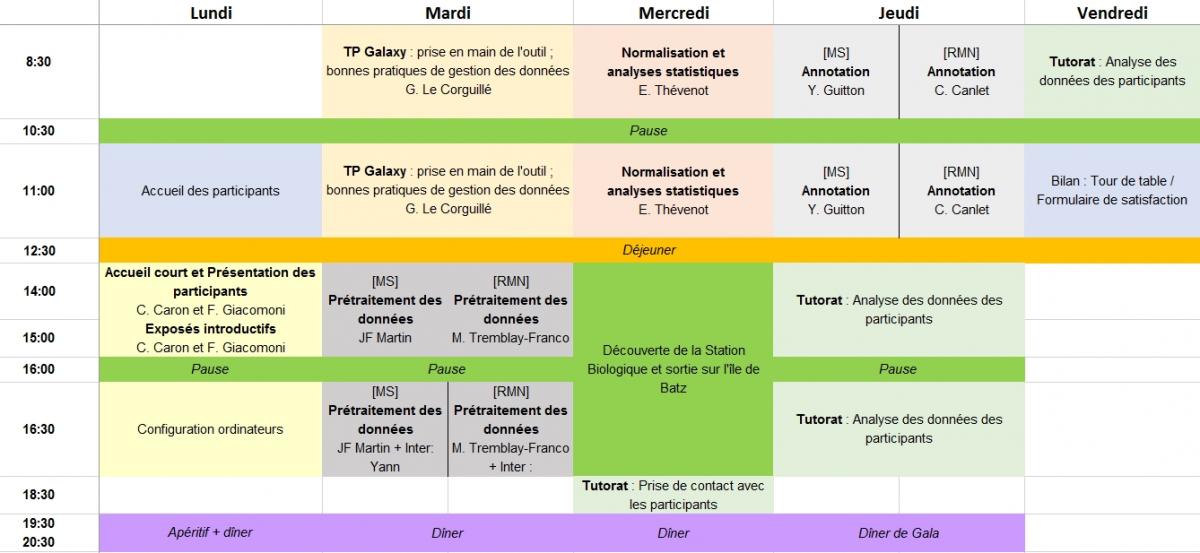
Lundi 21 septembre
11H00 - 12H30 : Accueil des participants
14H00 - 14H15 : Présentation de l'école
Intervenants : C. Caron, F. Giacomoni
14H15 - 15H15 : Tour de table
Présentation par les participants de leur cursus (2 minutes par stagiaire)
15H15 - 16H00 : Exposés introductifs
Intervenants : C. Caron, F. Giacomoni
- Présentation des infrastructures nationales
- IFB (C. Caron)
- MetaboHUB (F. Giacomoni)
- Exposé scientifique : Catherine Leblanc (UMR 8227)
- Interactions algues/herbivores : défense et signalisation chimique.
16H30 - 18H00 : Configuration des ordinateurs portables
Mardi 22 septembre
8H30 - 12H30 : TP Galaxy
Intervenants : G. Le Corguillé, M. Monsoor
-
Qu'est-ce que Galaxy ?
-
Découverte de l'interface
-
Les formats de données
-
Déposer ses données
-
Lancer un outil
-
Générer et éditer une chaine d'analyse
-
Partager ces données et sa chaine d'analyse
14H00 - 18H00 : Prétraitement des données
Intervenants : J.F. Martin pour le Groupe 1 (Yann, Alexis, Mélanie), M. Tremblay-Franco pour le groupe 2 (Cécile)
- Groupe 1 : MS
- Extraction des signaux (xcmsSet)
- Alignement entre échantillons (group)
- Correction des temps de rétention (Retcor)
- Production de la matrice de résultats (diffreport)
- Annotation des Isotopes et adduits (CAMERA)
- Correction de dérive analytique et d'effet "batch"
-
Groupe 2 : RMN
-
Bucketing et intégration
-
Normalisation
-
Mercredi 23 septembre
8H30 - 12H30 : Normalisation et analyses statistiques
Intervenants : E. Thévenot (Mélanie, Marie, Gildas)
- Formattage des données pour le post-processing (Mélanie, ~30 min)
- Analyses univariées (Marie, Gildas, ~1h15)
- Analyses multivariées : PCA, (O)PLS et (O)PLS-DA (Etienne, ~1h45).
14H00 - 18H00 : Découverte de la Station Biologique de Roscoff
- Découverte de le Station Biologique de Roscoff
- Sortie sur l'île de Batz
Jeudi 24 septembre
8H30 - 12H30 : Annotation
Intervenants : Y. Guitton (Alexis, Franck, Jean-François, Marion), C. Canlet (Rémi, Marie)
-
Groupe 1 : MS
- Le challenge de l'annotation : du m/z (du spectre) à la molécule.
- Interprétation des résultats CAMERA (les pcgrp),
- Utilisation avancée de CAMERA (les rules)
- Traitement d'exemples
- Utilisation des briques d'interrogation de banques sur bases m/z (HR2Formula, HMDB, MassBank ...)
- option 1 : Interrogation de banques sur base de spectres de masse - Golm pour la GCMS, MassBank pour GCMS/LCMS
- option 2 : ProbMetab et annotation basée sur un réseau métabolique de référence.
-
Groupe 2 : RMN
-
Challenge de l'annotation de spectres de mélanges
-
Etat des lieux des méthodes ou logiciels existants
-
Présentation de la méthode basée sur la comparaison avec des spectres de composés de référence
-
Estimation des déformations
-
Estimation des proportions
-
Traitement d'exemples
-
14H00 - 18H00 : Tutorat
Analyse des données des participants
Vendredi 25 septembre
8H30 - 10H30 : Tutorat
Analyse des données des participants
11H00 - 12H30 : Bilan de l'école
- Questionnaire en ligne
- Tour de table
Le prétraitement des données RMN concerne les étapes de bucketing et la normalisation.
Pour le prétraitement des données et l'annotation, deux sessions se dérouleront en parallèle de manière à couvrir spécifiquement les aspects MS et RMN.
Durant les sessions "tutorat", les participants seront accompagnés pour traiter leur propre jeu de données.